昨日紹介したAlphaFold DatabaseでのFoldseekの使い方をスクショを交えて説明してみます。
こちらをクリックしてみてください。https://alphafold.ebi.ac.uk/entry/P06620
人工雪を作るときの核になるバクテリアがもっているタンパク質InaZのサイトのurlで、クリックして開くのがこんなページです。
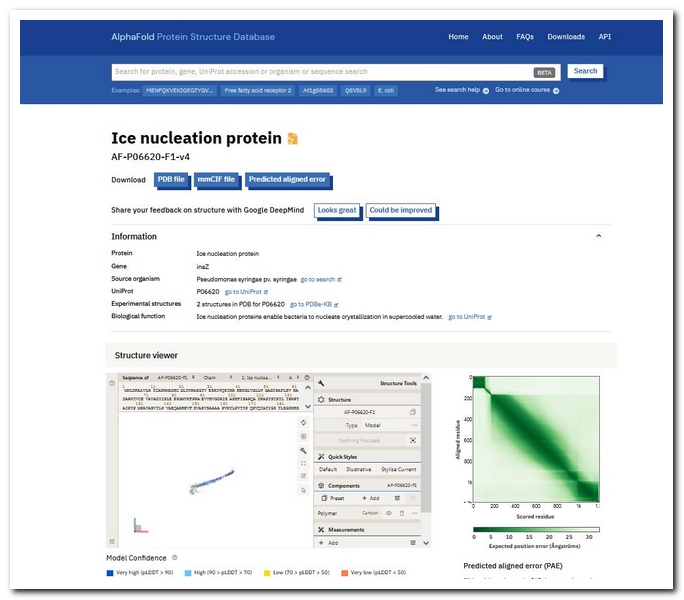
下の方に立体構造が3Dで表示されています。マウスで回転させたり拡大縮小表示したりできることを確認してください。
もっと下の方に画面をスクロールすると、Foldseekの結果が表示されているのがわかります。
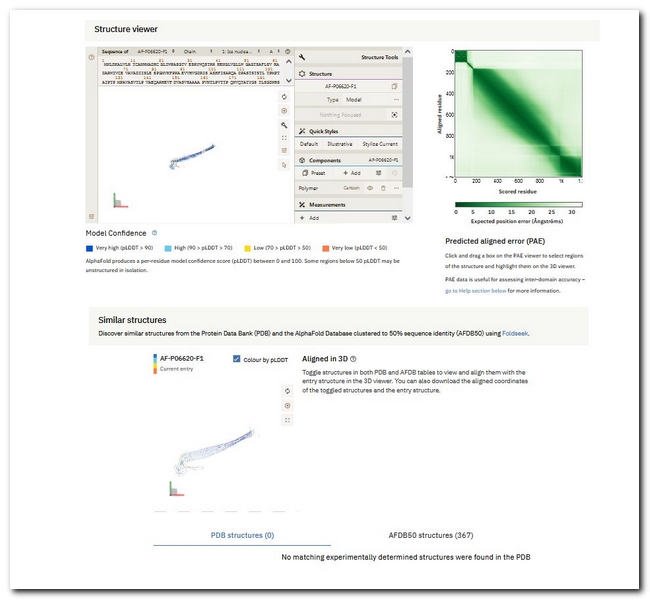
Similar structuresという部分を見てください。PDBに登録されているものでヒットは0です(PDB structures (0)と表示されている部分)が、右のAFDB50 structures (367)とある部分をクリックすると367個のヒットを見ることができます。
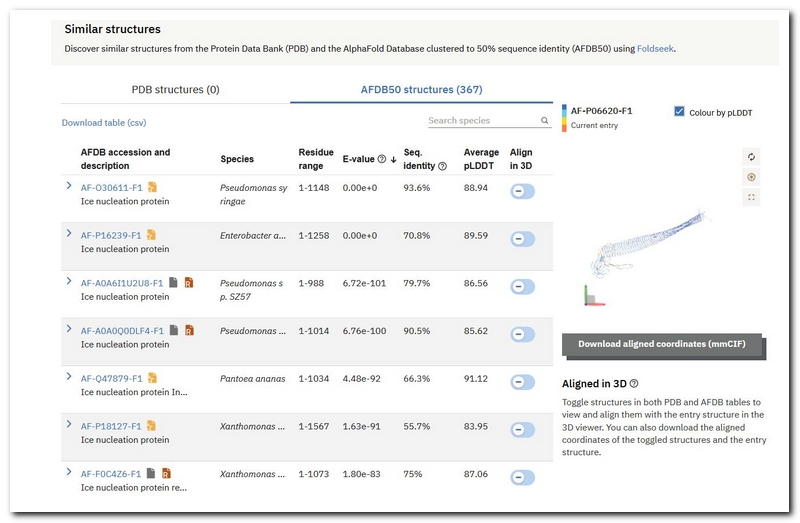
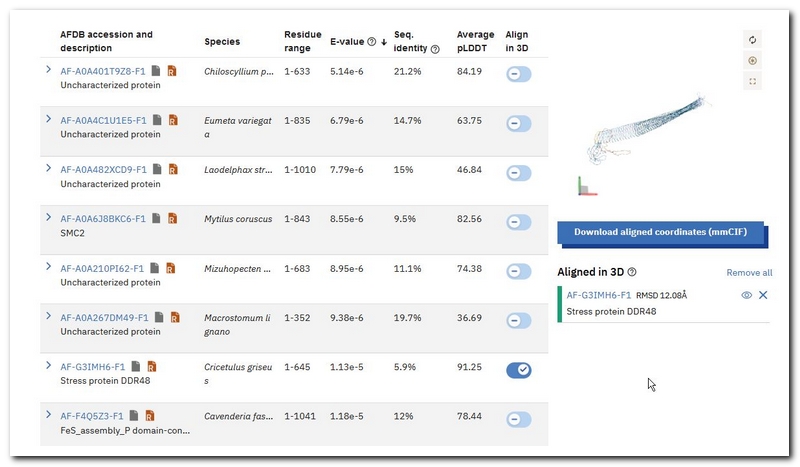
この図では、7個のヒットが表示されています。表になっていますが一番右の列Align in 3DがInaZとヒットしたタンパク質を重ねて立体表示するためのスイッチです。-になっているのは表示されていない状態を示します。クリックしてやるとクリックしたタンパク質がInaZの立体構造に重ねて表示されます。
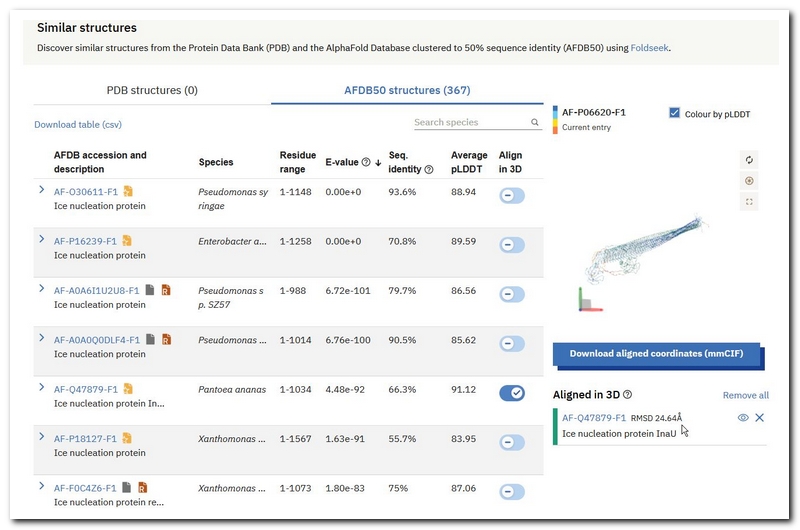
前の図と較べると、チェックが入っているタンパク質の立体構造が並べて表示されているのがわかります。複数の行でクリックすると、クリックしたタンパク質が全部アラインして表示されます。右下のAligned in 3Dの部分の下をみると、タンパク質のIDの右にRMSD (root mean square deviation)が表示されており、この例では、24.64Åとなっています。RMSDの値が低いほどよくアラインしていることを示します。
では昨日書いたストレスタンパク質をInaZにアライメントしてみましょう。
右上のバネのように巻いている部分にアラインしているのがわかります。RMSD値は低いですね。
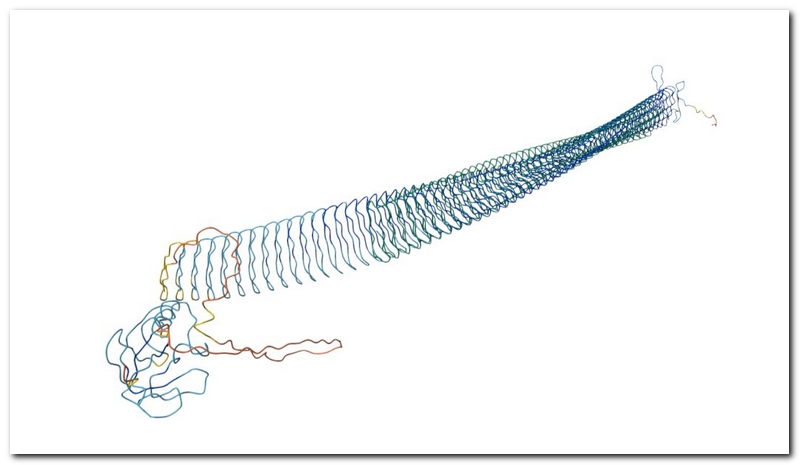
緑のバネのような部分がStress Proteinです。InaZはこの部分では青になっています。
もっと拡大して、一部分にカーソルを当ててみましょう。

矢印のカーソルの部分が赤く囲まれています。この部分の説明が右下にでています。マウスカーソルの矢印が当たっている部分のアミノ酸はイソロイシン(アミノ酸の略号がILE)で240番目のアミノ酸であることがわかります。
以上のような要領で、自分の好きなタンパク質を表示して遊んでみてください。もちろんアライメントの座標などもダウンロードすることができます。