2016.07.07 (木) 次世代シークエンサーの威力―DNase-Seq(ディーエヌエース シークと読みます)とATAC-seq (seqをセックと読む人もいますが英語ではシークと読むのが多いです): 七夕です。福岡は快晴 (7月分ブログ初日です) 。
最近活用されている次世代シークエンサーの威力を基幹教育の細胞生物学の講義で紹介しました。染色体の中で活発に機能しているDNAの部分はDNase I というDNAを切断する酵素で切れやすくなっていることがわかっています。そこで核をとりだして軽くDNase I 消化を行って活発に機能しているゲノム部分を消化した後、次世代シークエンサーで切れた部分の配列を同定すれば、その細胞で活発に働いているゲノムの部分の塩基配列がわかります。これをDNase-Seq解析と呼びます。たとえば幹細胞から分化する前と後でこの解析を行うと、幹細胞で発現していたタンパク質遺伝子や遺伝子制御にかかわる制御遺伝子のおよそ半分は、分化した細胞でも活発に機能して発現しており、新たに半分くらいの分化した細胞に特徴的な遺伝子が発現しているようです。このDNase-Seq解析は、
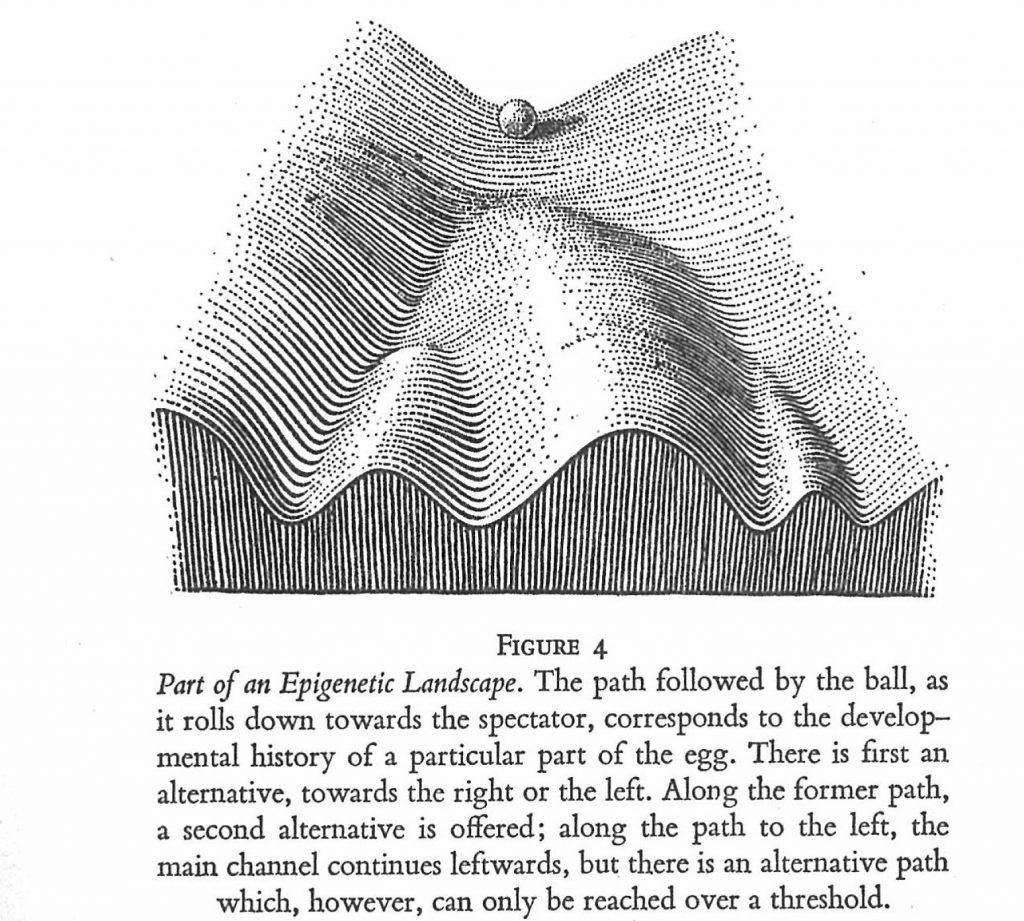
有名な20世紀の発生学者Waddingtonのepigenetic landscapeの実体を明らかにしつつあると考えられ、わくわくします。詳しくは オープンアクセスの論文Developmental Fate and Cellular Maturity Encoded in Human Regulatory DNA Landscapesをご覧ください。写真は九大図書館にあるWaddingtonの著書The Strategy of the Genes (1957)からスキャンしたものです。
 ヒトではタンパク質をコードする遺伝子は21,000個ほどあり、そのほかに4,0000個ほどのnon-coding RNAをコードする遺伝子( プロモーターをもちexonがあり、スプライシングをうけるといった普通の遺伝子の特徴を備えているもの)があるほか、100万個をこえるfocal smaller/less structured non-coding RNAsが同定されているようです(ビデオ)。
ヒトではタンパク質をコードする遺伝子は21,000個ほどあり、そのほかに4,0000個ほどのnon-coding RNAをコードする遺伝子( プロモーターをもちexonがあり、スプライシングをうけるといった普通の遺伝子の特徴を備えているもの)があるほか、100万個をこえるfocal smaller/less structured non-coding RNAsが同定されているようです(ビデオ)。
DNase-seqのほかにも、感度を大幅に上げた方法(1細胞でも解析可能らしい)として、Tn5 のtransposaseという酵素を利用するATAC(Assay of transposase Accessible Chromatin)-Seqという方法(ビデオ)も知られています。これはトランスポゾンが、開いている染色体部分に飛び込むメカニズムを利用する方法です。トランスポゾンは自身をゲノムに組み込むときに、宿主側のゲノムDNAに自身のDNA配列を結合させる(ライゲーションさせる)活性をもつ酵素transposaseをもっています。高い活性のtransposaseを遺伝子改変によって作成し、この発現させたtransposaseといっしょにPCR増幅用のプライマーを入れることで、開いているクロマチン部分にtransposaseによってPCR用のプライマー配列を結合させることができます(transposaseのligation活性を利用)。こうして開いているクロマチン部分だけに増幅用プライマーを導入した後、挿入したプライマーを利用してPCRで増幅すれば、開いていたクロマチン部分が増幅されて、あとは次世代シークエンサーで増幅された配列を読み取り、どんな配列の部分が開いていたかが網羅的にきまるという仕組みです。 (2019.08.21追記:またRNA-Seqも有用な技術として広く活用されており、一細胞のレベルでのRNA発現パターンの解析も今や可能になっています。こちらの記事を参照してください。)
このような新しい手法の開発で、発生のステップごとにどのように遺伝子の発現調節が変化するかや、病気の発症段階をおっての遺伝子発現調節の変化が解明され、遺伝子制御ネットワークの全貌(レギュロームという名前も提唱されています)が解明されています。まさに生物学の革命を私たちは目にしているのです。